In dem Film „Jurassic Park“ extrahierten Wissenschaftler DNA, die Millionen von Jahren in Bernstein aufbewahrt worden war, und nutzten sie, um eine Population längst ausgestorbener Dinosaurier zu erschaffen.
Inspiriert von diesem Film haben MIT-Forscher ein glasartiges, bernsteinähnliches Polymer entwickelt, das für die Langzeitspeicherung von DNA verwendet werden kann, sei es für ganze menschliche Genome oder digitale Dateien wie Fotos.
Aktuelle Methoden brauchen zu viel Energie
Die meisten derzeitigen Methoden zur Speicherung von DNA erfordern Gefriertemperaturen, verbrauchen also viel Energie und sind in vielen Teilen der Welt nicht praktikabel. Im Gegensatz dazu kann das neue bernsteinähnliche Polymer die DNA bei Raumtemperatur speichern und gleichzeitig die Moleküle vor Schäden durch Hitze oder Wasser schützen.
Die Forscher haben gezeigt, dass sie mit diesem Polymer sowohl DNA-Sequenzen, welche die Titelmusik von Jurassic Park kodieren, als auch ein ganzes menschliches Genom speichern können. Sie zeigten auch, dass die DNA leicht aus dem Polymer entfernt werden kann, ohne sie zu beschädigen.
„Das Einfrieren von DNA ist die beste Methode, um sie zu konservieren, aber es ist sehr teuer und nicht skalierbar“, sagt James Banal, ein ehemaliger Postdoc am MIT. „Ich glaube, dass unsere neue Konservierungsmethode eine Technologie sein wird, die die Zukunft der Speicherung digitaler Informationen auf der DNA bestimmen wird.“
Banal und Jeremiah Johnson, der A. Thomas Geurtin Professor für Chemie am MIT, sind die Hauptautoren der Studie. Die ehemalige MIT-Postdoc Elizabeth Prince und MIT-Postdoc Ho Fung Cheng sind die Hauptautoren der Studie.
DNA einfangen
Die DNS, ein sehr stabiles Molekül, eignet sich gut für die Speicherung großer Mengen an Informationen, einschließlich digitaler Daten. Digitale Speichersysteme kodieren Text, Fotos und andere Informationen als eine Reihe von 0en und 1en. Dieselben Informationen können in der DNA mit Hilfe der vier Nukleotide kodiert werden, die den genetischen Code bilden: A, T, G und C. G und C könnten zum Beispiel für die 0 stehen, während A und T die 1 darstellen.
Die DNA bietet eine Möglichkeit, diese digitalen Informationen in sehr hoher Dichte zu speichern: Theoretisch könnte eine Kaffeetasse voller DNA alle Daten der Welt speichern. Außerdem ist die DNA sehr stabil und relativ einfach zu synthetisieren und zu sequenzieren.
Im Jahr 2021 entwickelten Banal und sein Postdoc-Berater Mark Bathe, ein MIT-Professor für Biotechnologie, eine Methode zur Speicherung von DNA in Siliziumdioxidpartikeln, die mit Markierungen versehen werden konnten, die den Inhalt der Partikel offenbarten. Diese Arbeit führte zu einer Ausgründung namens Cache DNA.
Ein Nachteil dieses Speichersystems ist, dass es mehrere Tage dauert, bis die DNA in die Silikapartikel eingebettet ist. Außerdem wird zum Entfernen der DNA aus den Partikeln Flusssäure benötigt, die für die Arbeiter, die mit der DNA arbeiten, gefährlich sein kann.
Um alternative Speichermaterialien zu entwickeln, begann Banal mit Johnson und Mitgliedern seines Labors zu arbeiten. Ihre Idee war es, eine Art von Polymer zu verwenden, das als abbaubarer Duroplast bekannt ist und aus Polymeren besteht, die beim Erhitzen einen Feststoff bilden. Das Material enthält auch spaltbare Verbindungen, die leicht gebrochen werden können, so dass das Polymer auf kontrollierte Weise abgebaut werden kann.
„Bei diesen dekonstruierbaren Duroplasten können wir je nachdem, welche spaltbaren Bindungen wir in sie einbauen, wählen, wie wir sie abbauen wollen“, sagt Johnson.
Die T-REX-Methode
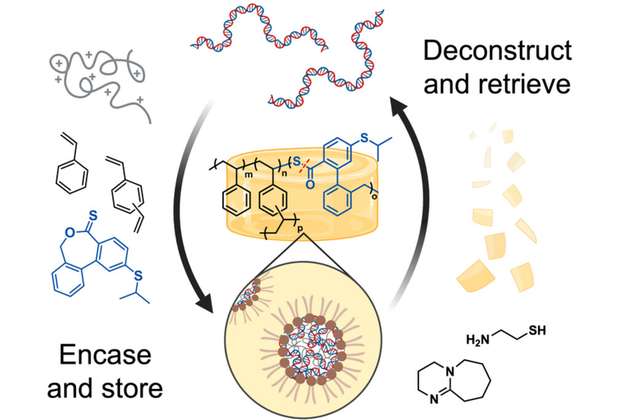
Für dieses Projekt entschieden sich die Forscher, ihr duroplastisches Polymer aus Styrol und einem Vernetzer herzustellen, die zusammen einen bernsteinähnlichen Duroplast namens vernetztes Polystyrol bilden. Dieser Duroplast ist außerdem sehr hydrophob, so dass er verhindern kann, dass Feuchtigkeit eindringt und die DNA beschädigt. Um den Duroplast abbaubar zu machen, werden die Styrolmonomere und Vernetzer mit Monomeren namens Thionolactonen copolymerisiert. Diese Verbindungen können durch Behandlung mit einem Molekül namens Cysteamin gebrochen werden.
Da Styrol so hydrophob ist, mussten sich die Forscher etwas einfallen lassen, um die DNA – ein hydrophiles, negativ geladenes Molekül – in das Styrol zu locken.
Dazu identifizierten sie eine Kombination aus drei Monomeren, die sie in Polymere verwandeln konnten, die die DNA auflösen, indem sie ihr helfen, mit Styrol zu interagieren. Jedes der Monomere hat unterschiedliche Eigenschaften, die zusammenwirken, um die DNA aus dem Wasser und in das Styrol zu bringen. Dort bildet die DNA kugelförmige Komplexe, mit geladener DNA in der Mitte und hydrophoben Gruppen, die eine äußere Schicht bilden, die mit dem Styrol interagiert. Beim Erhitzen wird diese Lösung zu einem festen glasartigen Block, in den die DNA-Komplexe eingebettet sind.
Die Forscher tauften ihre Methode T-REX (Thermoset-REinforced Xeropreservation). Der Prozess der Einbettung der DNA in das Polymernetzwerk dauert einige Stunden, könnte aber bei weiterer Optimierung kürzer werden, sagen die Forscher.
Um die DNA freizusetzen, fügen die Forscher zunächst Cysteamin hinzu, das die Bindungen spaltet, die den Polystyrol-Duroplast zusammenhalten, und ihn in kleinere Stücke zerlegt. Anschließend kann ein Detergens namens SDS hinzugefügt werden, um die DNA aus dem Polystyrol zu entfernen, ohne es zu beschädigen.
Speicherung von Informationen
Mit Hilfe dieser Polymere konnten die Forscher zeigen, dass sie DNA unterschiedlicher Länge einkapseln können, von einigen zehn Nukleotiden bis hin zum gesamten menschlichen Genom (mehr als 50.000 Basenpaare). Sie waren in der Lage, DNA zu speichern, die die Emanzipationsproklamation und das MIT-Logo sowie die Titelmusik von „Jurassic Park“ enthält.
Nach der Speicherung der DNA und ihrer anschließenden Entnahme sequenzierten die Forscher sie und stellten fest, dass keine Fehler aufgetreten waren, was ein kritisches Merkmal jedes digitalen Datenspeichersystems ist.
Die Forscher wiesen auch nach, dass das duroplastische Polymer die DNA vor Temperaturen von bis zu 75 °C schützen kann. Sie arbeiten nun daran, den Herstellungsprozess der Polymere zu rationalisieren und sie zu Kapseln für die Langzeitspeicherung zu formen.
Cache DNA, ein von Banal und Bathe gegründetes Unternehmen, bei dem Johnson Mitglied des wissenschaftlichen Beirats ist, arbeitet nun an der Weiterentwicklung der DNA-Speichertechnologie. Die erste Anwendung, die sie sich vorstellen können, ist die Speicherung von Genomen für die personalisierte Medizin, und sie gehen auch davon aus, dass diese gespeicherten Genome weiter analysiert werden könnten, wenn in Zukunft bessere Technologien entwickelt werden.
„Die Idee ist, warum bewahren wir nicht die Hauptaufzeichnung des Lebens für immer?“ sagt Banal. „In zehn oder 20 Jahren, wenn die Technologie viel weiter fortgeschritten ist, als wir es uns heute vorstellen können, könnten wir mehr und mehr lernen. Wir stehen noch ganz am Anfang, wenn es darum geht, das Genom zu verstehen und wie es mit Krankheiten zusammenhängt.“
Dieser Artikel wurde mit Deepl aus dem Englischen übersetzt.